피플
[남궁석의 신약연구史]X선 결정학과 단백질 구조규명
남궁석 SLMS(Secret Lab of Mad Scientist) 대표
[단백질 구조부터 신약까지①]X선 결정학의 태동..회절 패턴부터 구조까지
현대 생물학은 근원적으로 생명을 구성하는 분자수준에서 생명의 원리를 탐구하며, 여기의 핵심에는 생명체 내에서 수많은 기능을 직접 수행하는 ‘생명의 부품'인 단백질과 같은 생체 고분자의 역할이 중심이 되어있다. 특히 이들이 원자수준에서 어떤 구조를 가지고 있고, 이러한 생체 고분자의 구조가 어떻게 이들의 생물에서의 기능을 수행하는지를 이해하는 것은 현대 생명과학의 핵심적인 부분인 셈이다.
이제 수만 종류의 단백질의 입체구조가 규명되어 있고, 이러한 정보에 근거하여 알파폴드2 등의 컴퓨터 알고리즘이 아직 실험적으로 입체구조가 규명되어 있지않은 어떤 단백질도 단백질의 진화정보에 근거하여 비교적 정확하게 구조를 예측해 낼 수 있는 상황에서 어떤 과정을 통해 단백질 구조가 규명되게 되었는지를 짚어보는 것은 큰 의미가 있다. 또한 오늘날의 신약개발에 있어서 단백질과 약물의 결합구조가 차지하는 중요성을 생각하며 보면, 단백질 구조가 규명된 역사를 아는 것은 오늘날의 신약개발을 이해하는 중요한 열쇠이기도 하다.
앞으로 수 회의 연재를 통하여 X선 결정학에 의해 단백질의 3차원 구조가 규명되는 과정, 그리고 단백질 구조의 규명이 어떻게 단백질의 기능의 이해와 나아가서 생명 현상의 원리의 이해로 진행되는 과정, 그리고 어떻게 단백질 구조가 신약개발 과정을 바꾸게 되었는지, 그리고 21세기에 들어 단백질 구조 생물학에서 새롭게 혁신을 불러일으키고 있는 초저온 전자현미경에 의한 단백질 구조 규명의 과정까지 다루어 보도록 하겠다.
X선 결정학의 태동
1895년 독일의 물리학자 빌헬름 뢴트겐은 파장이 가시광선보다 훨씬 짧은(10-0.01nm, 가시광선은 400-700nm) 전자기파를 발견했고, 이 전자기파는 투과력이 매우 강하여 사람의 몸을 투과하여 뼈를 관찰할 수 있었고, 비침습적으로 몸의 내부를 투과해 볼 수 있는 성질은 이후의 의료에 엄청난 영향을 미쳤다. 이 전자기파는 ‘뢴트겐선', 혹은 ‘X선' 으로 불리게 되었다.
기존의 가시광선에서는 볼 수 없었던 높은 에너지를 가진 전자기파인 X선은 곧 여러가지 흥미있는 현상을 유도한다는 것을 알게 되었다. 1912년 독일의 물리학자 막스 폰 라우에(Max Von Laue)는 X선을 황산구리의 결정에 노출시키면 X선이 특정한 패턴을 나타내는 것을 발견하였다. 이것은 결정 내의 원자구조가 규칙적으로 배열되어 있고, 결정 내의 원자에 X선의 파동이 접촉하면 간섭을 일으키기 때문이었다. 이러한 현상을 '회절(Diffraction)'이라고 부른다.
1913년 영국의 로렌스 브래그(Lawrence Bragg)와 헨리 브레그(Henry Bragg) 부자는 결정에서 X선이 회절되는 조건을 규명하였다.[1] 결정은 주기적인 구조를 가지고 있는 물질이고, X선을 다양한 각도에서 비춰주면 어떤 각도에서는 강한 회절이 일어나지만, 어떤 각도에서는 회절이 일어나지 않는다. 이러한 현상이 일어나는 것은 결정을 구성하는 물질의 원자에 X선이 충돌하여 파장의 방향이 바뀔 때, 어떤 경우에는 파장이 중첩되어 강해지기도 하고, 어떤 경우에는 약해지기 때문에 일어나는 현상이다. 브래그는 격자에서 회절되어 나온 X선이 나타나는 조건을 수식으로 확립하였고 이 수식은 ‘브랙의 법칙(Bragg’s Law)'으로 알려졌다. 이를 이용하여 여러가지 무기물의 결정 내에서 원자가 어떻게 배열되어 있는지를 알 수 있게 되었다. 결정 격자의 X선 회절 패턴을 분석함으로써 결정 내의 원자들의 배열에 대한 정보를 알 수 있다는 것이다. 이것이 바로 무기물, 유기 화합물부터 단백질까지 수많은 화학물질의 구조분석을 가능케 한 기술인 X선 결정학의 시작이다.
처음에는 단순한 무기물 결정구조를 밝히는 것으로 시작되었던 X선 결정학은 좀 더 복잡한 탄소 화합물의 구조를 결정하는 방법으로 사용되기 시작한다. 1925년 브래그의 제자였던 존 데스몬드 버널(John Desmond Bernal)은 흑연(Graphite) 결정을 X선 회절로 분석하여 흑연이 탄소가 육각형 형태로 이루어진 격자구조로 되어 있다는 것을 밝혔다. 그 이후 X선 결정학은 여러가지 유기 화합물의 구조를 밝히는 도구로 사용된다. 이러한 발전은 1947년 항생제 페니실린(Penicillin), 1952년 비타민 B12의 구조와 같이 복잡한 유기 화합물의 구조 규명으로 이어진다.
그러나 가장 복잡한 탄소 화합물은 바로 단백질이고, X선 결정학은 단백질의 구조를 밝히는 핵심적인 도구로 사용되게 된다. 그러나 X선 결정학에 의해서 단백질의 구조를 알아보는 과정에 대해서 이야기하기 전에 일단 ‘단백질은 고정된 구조를 가지고 있다'는 것이 어떻게 받아들여지게 되었는지의 과정부터 알아보도록 하자.
단백질의 결정화
오늘날 많은 단백질들이 단백질마다 다른 고유한 3차원 구조를 가지고 있고, 이러한 단백질의 3차원 구조가 단백질의 고유 기능을 결정한다는 것은 상식처럼 알려져 있다. 그러나 오늘날의 많은 ‘상식적 지식'과 마찬가지로 이것도 역시 20세기 초에는 전혀 상식이 아니었다.
물론 20세기 초반에도 이미 단백질이 수 천에서 수 만에 이르는 분자량을 지닌 고분자 물질이라는 것은 알려져 있었고, 아미노산이 중합되어 이루어진 폴리펩타이드라는 것 역시 알려져 있었다. 그리고 생체 내에서 여러가지 화학반응을 촉매하는 ‘효소' 역시 단백질이라는 것이 알려지고 있던 참이었다. 그러나 그 당시 학계의 주된 견해는 단백질은 특정한 형태를 가지지 않은 무정형의 고분자이며, 효소의 촉매반응은 효소에 연결되어 있는 보결원자단(Prosthetic group, 헤모글로빈에서 산소를 결합하고 있는 헴(Heme)이 그 좋은 예이다)에 의해서 이루어지고, 단백질은 그저 이러한 보결원자단을 지지하는 담체 정도의 의미라는 것이었다. 특히 이러한 견해는 20세기 초중반까지 생화학계의 주류를 형성하던 독일을 중심으로하는 유럽학계에서 지배적이었다.
(물론 오늘날의 우리는 단백질 중에는 뚜렷한 3차원 구조를 가지고 있는 것도 있지만, 반면에 뚜렷한 3차 구조를 가지지 않는 무정형의 단백질도 꽤 많이 존재하며, 어떤 단백질은 뚜렷한 3차 구조를 가진 도메인과 무정형 도메인을 동시에 가지고 있다는 것도 알고있다. 그러나 이러한 현대의 상식은 일단 많은 단백질, 특히 세포 내에서 효소와 같은 특정한 화학반응을 촉매하는 단백질들은 고정된 3차원적 구조를 가지고 있다는 것이 밝혀진 이후에 정립된 것이다)
그러나 단백질이 뚜렷한 3차원 형태를 가지고 있다는 것을 암시하는 증거가 속속 출현하였다. 먼저 1926년 미국의 생화학자 제임스 섬너(James B Sumner, 1887-1955)다. 코넬 대학의 교수였던 섬너는 작두콩(Jack Bean)에서 요소를 분해하는 요소분해효소(Urease)를 정제하고 이를 결정화하였다.[2] 그렇다면 결정을 형성한다는 것이 어떻게 단백질이 무정형의 고분자가 아니라는 증거가 될까? 결정은 어떤 물질이 원자수준에서 격자를 형성하여 만들어지고, 그 격자를 형성하는 구성물은 화학적으로 동일하다. 가령 소금 결정은 소듐(Na+)과 염소(Cl-) 이온이 동일한 구조로 배열되어 있다. 즉 단백질이 결정을 형성할 수 있다는 것은 단백질 분자가 동일한 형태로 결정을 구성하는 격자에 배열되어 있다는 것이며, 만약 단백질 분자가 일정한 형태가 없는 무정형으로 존재한다면 결정을 형성할 수 없을 것이다.
이렇게 단백질, 적어도 결정화된 단백질이 뚜렷한 3차원 형태를 가지고 있다는 것은 이렇게 형성된 단백질 결정을 X선에 쪼이면 회절현상이 일어난다는 것을 발견한 이후 더욱 확실해졌다. 1934년 버널과 도로시 크로포드(Dorothy Croford, 이후 결혼 후 도로시 크로포드 호지킨 Dorothy Croford Hodgkin이라는 이름으로 알려졌으며, 페니실린, 비타민 B12 및 인슐린의 구조를 규명하여 1965년 노벨 화학상을 수상한다)는 단백질 분해효소인 펩신(Pepsin)의 결정을 X선에 노출시키면, 이 결정은 회절 패턴을 만들어 낸다는 것을 알게 되었다.[3] 단백질 결정이 X선을 회절시킨다는 것을 확인하기까지는 어느 정도의 시행착오가 있었는데, 처음에는 다른 무기물 결정과 마찬가지로 결정을 건조시킨 다음 X선을 쬐었을 때는 아무런 회절패턴이 나타나지 않았다. 그러나 단백질 결정 역시 다른 세포 내의 구성물과 마찬가지로 물의 함량이 70% 이상 차지한다는 것을 알게 된 이후, 단백질 결정을 액체상태에서 유지하기 위하여 가는 유리 대롱에 담아 액체 내에서 유지한 상태에서 X선을 쬐었을때는 무수하게 많은 회절패턴이 나타났다. 이러한 회절패턴은 단백질의 종류 및 결정의 종류에 따라서 달라졌고, 이 회절패턴은 단백질의 구조를 반영하고 있는 셈이었다.
그러나 어떻게 이러한 회절패턴을 통하여 단백질의 구조를 유추할 수 있을까?
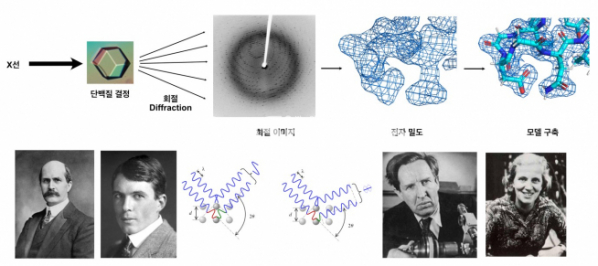
▲그림 1. (상) X선 결정학에 의한 단백질 구조 규명. 단일 결정으로 결정화된 물질에 X선을 가하면, 결정화된 물질의 원자에 X선이 충돌한 후 산란되고, 산란되는 X선 파동은 결정 안의 원자구조에 따라서 서로 상쇄 및 간섭을 일으키는 회절(Diffraction)현상을 일으킨다. 이 회절 패턴은 결정화된 물질의 원자 구조에 대한 정보를 담고 있으며, 회절 패턴을 분석하여 결정 내의 물질의 원자 구조를 전자밀도(Electron Density) 형태로 얻어낸 후, 이에 기반하여 원자 수준의 분자구조를 결정하는 것이 바로 X선 결정학의 원리이다. (하) X선 결정학의 기여자들. 로렌스 브래그(Lawrence Bragg)와 헨리 브레그(Henry Bragg)는 결정화된 물질에 X선을 쪼여서 회절과 상쇄가 일어나는 규칙인 ‘브렉의 법칙(Bragg’s Law)’을 발견하고, 이를 이용하여 소금 결정의 원자구조를 처음으로 밝혔다. 한편 존 데스몬드 버널(John Desmond Bernal)과 도로시 크로포드(Dorothy Crowford)는 단백질 결정의 회절 패턴을 처음으로 관찰하여, 결정 내의 단백질 원자가 모두 고정된 형태의 구조를 가지고 있음을 관찰했다.
회절 패턴으로부터 구조까지
오스트리아 출신의 막스 퍼루츠(Max Perutz, 1914-2002)는 1936년 캠브리지대 캐번디시 연구소로 유학와서 버널의 지도를 받으며 단백질 X선 결정학의 연구를 시작한다. 퍼루츠가 연구 대상으로 삼은 단백질은 혈액 내에서 산소를 결합하는 단백질인 헤모글로빈이었다. 1938년 퍼루츠는 버널과 함께 헤모글로빈의 결정의 X선 회절결과를 보고한다.[4] 그의 결정은 X선을 매우 잘 회절하였고, 최대 2 옹스트롬(10^-10m 또는 0.1nm) 간격의 회절신호도 포착되었다. 이것은 단백질 결정에서 나오는 회절신호는 단백질을 구성하는 개별적인 원자에서 나오는 신호를 구별할 정도로 고해상도의 정보였고, 단백질 구조로부터 유래된 회절 신호를 분석하면 단백질의 구조를 원자 수준에서 파악할 수 있다는 것을 암시하는 결과였다.[5]
그러나 문제는 어떻게 이 신호를 해석할 것인가? 단백질 결정에서 나오는 X선 회절은 X선 필름에 감광되어 저장하며, 이 정보로 알 수 있는 것은 원자로부터 나온 X선의 신호의 세기였다. 그러나 이를 이용하여 X선을 회절하는 원자의 형태(단백질의 구조)를 알기 위해서는 회절신호의 세기 (파동에서의 ‘진폭') 만을 아는 것으로는 불충분했다. 각각의 단백질 원자에 맞고 튕겨나오는 X선 파동의 진폭뿐만 아니라, 각각의 위상(Phase)을 알아야 단백질 원자의 위치의 재구성이 가능했다.
좀 더 직관적인 비유(반드시 정확하지는 않을 수 있는)로 설명한다면 X선 회절신호를 이용하여 단백질의 원자구조를 파악하는 것은 빛의 그림자를 이용하여 물체의 완전한 3차원적인 구조를 파악하려는 것과 비슷하다. 그림자는 물체의 윤곽이나 전반적인 크기는 알려주지만, 구체적으로 그 물체가 가지고 있는 모든 특징을 알려주지는 않는다. 360도 각도로 빛을 쬐어 생성된 사람의 그림자를 아무리 열심히 살펴봐도 사람의 얼굴이나 입고 있는 옷에 대한 정보를 알기 어려운 것과 마찬가지다.
퍼루츠가 직면한 문제도 바로 이러한 문제였다. 단백질 결정에서 나오는 X선 회절로는 회절 파동의 세기는 알 수 있지만, 생성된 파동에서 파동을 형성한 원자가 어떤 위치에 있는지(위상) 알 수는 없다. 즉 회절 데이터만 가지고는 단백질의 원자구조를 풀기는 불충분하다는 것이다. 회절 데이터에는 분명히 단백질의 구조에 대한 정보가 들어있지만, 구조를 실제로 풀기 위해서는 마치 암호를 풀기 위한 열쇠인 위상 정보가 필요하다는 것이다. 이러한 문제는 쉽게 풀리지 않았다.
그러던 와중 2차 세계대전이 발발하고 당시 영국에 있던 퍼루츠는 다른 대부분의 과학자처럼 자신이 원래 연구하던 분야를 떠나서, 전쟁 수행에 필요한 다른 연구를 수행하였고(퍼루츠는 얼음을 이용하여 항공모함을 만드는 ‘하박국 계획'이라는 군사 프로젝트에 관여하였다) 연구는 진행되지 않고 멈춰 있었다.
2차 세계대전이 끝난 이후에야 퍼루츠는 결정학 연구에 복귀하였고, 1946년 퍼루츠의 연구에 존 켄드류(John Kendrew, 1917-1997)가 합류하였고, 켄드류는 헤모글로빈과 같이 산소를 결합하는 단백질이지만, 근육에서 산소를 결합하는 역할을 하며, 좀 더 간단한 단백질인 미오글로빈의 구조의 연구를 시작하였다. 1948년에는 물리학 전공의 대학원생인 프랜시스 크릭(Francis Crick, 1916-2004)이 합류하였다.(나중에 프랜시스 크릭은 미국 출신의 방문 학자인 제임스 왓슨과 함께 DNA의 이중 나선 모델을 발표하여 유명해졌다. 그러나 프랜시스 크릭의 공식적인 박사학위 주제는 바로 단백질의 3차원 구조를 규명하는 방법을 찾는 것이었다) 그러나 여전히 X선 회절의 위상 정보를 구하여 회절 정보로부터 단백질의 3차원 구조를 얻는 것에는 큰 진척이 없었다.
1954년에 되어서야 이러한 문제의 실마리가 풀렸다. 이들은 단백질 결정에 수은과 같은 중금속을 넣었을 때, 단백질의 특정한 위치에 수은이 결합하고, 이로 인해 회절 패턴의 극히 일부가 변하는 것이 발견되었다.[6] 중금속을 넣지 않고 얻은 결정의 회절패턴과 비교하여, 중금속의 결정 내에서의 위치를 결정할 수 있었고, 이를 통하여 단백질 원자의 위상을 어느 정도 계산할 수 있었다. 단백질 원자로부터 나오는 회절의 진폭과 위상을 알게 되면, 단백질을 구성하는 전자의 밀도를 계산할 수 있게 되고, 이렇게 얻어진 전자밀도에 기반하여 단백질의 구조 모델을 만들 수 있게 된다. 오늘날은 이러한 모든 과정을 컴퓨터로 수행 가능하지만, 이전에는 모두 손으로 하는 계산과 물리적인 원자 모델을 직접 함으로써 이루어졌고, 이 과정은 몇 년씩 걸리는 지난한 일이었다.
1958년 켄드류는 미오글로빈 전자 밀도 계산을 통해 얻어진 대략적인 미오글로빈의 구조를 제시하였다.[7] 이 결과는 아직 정확한 원자의 위치를 제시할 만큼 고해상도의 구조가 아니었고, 단백질의 윤곽 정도만을 구분할 수 있을 정도였다. 완전히 원자의 위치까지 구별할 수준의 구조는 1.5 옹스트롬까지 식별할 수 있는 결정 회절 데이터가 얻어진 이후인 1961년에 드디어 발표되었다.[8] 미오글로빈을 구성하고 있는 2,600개의 원자의 3차원 위치가 정확히 규명되었고, 이를 구성하고 있는 아미노산들과 산소를 결합하는 보결 원자단인 헴(Heme)의 위치까지 명백히 알려지게 되었다. 이것은 생명의 원초적인 분자인 단백질을 원자 수준에서 관찰하게 된 최초의 사건이었다.
켄드류가 미오글로빈의 구조를 규명하는 와중에 퍼루츠는 헤모글로빈의 구조 결정을 계속 진행하고 있었다. 미오글로빈과 헤모글로빈은 산소에 결합하는 단백질이라는 공통점을 가지고 있었지만 미오글로빈은 단백질 가닥 1개로 구성된 비교적 간단한 단백질이지만 헤모글로빈은 4가닥의 단백질(알파 서브유니트 2개, 베타 서브유니트 2개)이 모여서 만들어지므로 좀 더 큰 단백질이고, 따라서 구조 결정에도 좀 더 많은 시간이 요구되었다. 1960년 5.5 옹스트롬 정도의 해상도를 가진 최초의 헤모글로빈 모델이 만들어졌다.[9] 헤모글로빈을 구성하고 있는 4가닥의 단백질은 미오글로빈과 마찬가지로 하나의 헴을 결합하고 있었다.
이후 산소를 포함하고 있는 헤모글로빈과 산소가 없는 헤모글로빈의 구조가 각각 따로 결정되었고, 헤모글로빈 단백질은 산소의 결합 여부에 따라서 미세하게 구조가 변한다는 것이 발견되었다. 헤모글로빈이라는 산소를 결합하는 단백질은 산소 결합여부에 따라서 구조가 미세하게 변하는 ‘나노머신'과 같은 존재라는 것이다. 단백질의 3차원 원자구조를 알게 됨으로써, 생명체의 기본적인 부품인 단백질이 어떤 역할을 하는지를 파악하는 학문이 비로소 태동되기 시작한 것이다. 즉, 오늘날 ‘구조생물학(Structural Biology)'이라고 불리는 학문의 태동은 1960년대에 미오글로빈과 헤모글로빈의 구조가 규명된 이후 비로소 시작된 셈이다.
퍼루츠와 켄드류는 1962년 그들의 단백질 구조에 대한 연구에 대한 공헌으로 노벨 화학상을 수상하였다. 공교롭게도 1962년의 노벨 생리 의학상은 DNA의 이중 나선 구조 모델을 제시한 공로로 프랜시스 크릭, 제임스 왓슨, 모리스 윌킨스가 수상하였다. 결국 1962년의 노벨 화학상 및 생리의학상은 단백질과 DNA 의 구조의 규명에 관련된 사람들에게 주어졌고, 이렇게 생명 현상의 근본이 되는 물질의 원자 수준의 분자 구조의 규명의 시작은 생명과학을 서서히 변화시키기 시작하였다.
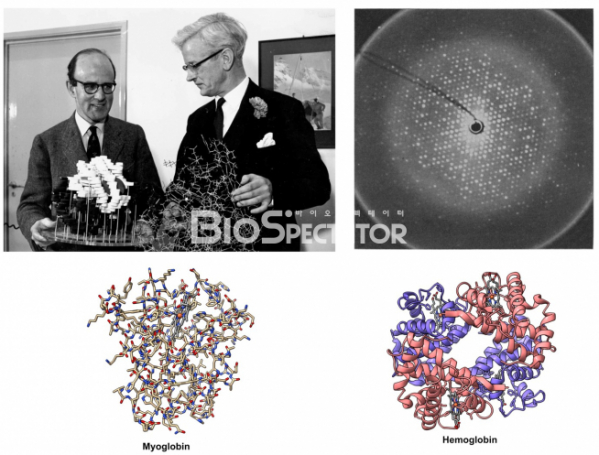
▲그림 2. X선 결정학에 의해 최초의 단백질 구조를 규명한 막스 퍼루츠(좌)와 존 켄드류(우). 이들은 각각 헤모글로빈과 미오글로빈이라는 단백질의 구조를 X선 결정학에 규명했다. 헤모글로빈의 X선 회절(우상단) 사진. 미오글로빈은 근육 내에서 산소를 결합하는 성질을 가지며, 헤모글로빈은 혈액 내에서 산소를 결합하여 운반하는 역할을 한다. 미오글로빈이 단백질 가닥 1개로 구성된 비교적 간단한 구조인 데 반하여 헤모글로빈은 4개의 단백질 가닥으로 구성된 보다 복잡한 단백질이다.
다음 연재에서는 헤모글로빈과 미오글로빈의 구조 규명으로 시작된 구조생물학이 어떻게 다양한 생체 고분자의 구조를 밝혀내어 우리의 생명에 대한 이해를 심화시켰는지를 알아보도록 하자.
참고문헌
1. Bragg, W. H., & Bragg, W. L. (1913). The reflection of X-rays by crystals. Proceedings of the Royal Society of London. Series A, Containing Papers of a Mathematical and Physical Character, 88(605), 428-438.
2. Sumner, J. B. (1926). The isolation and crystallization of the enzyme urease. Preliminary paper. J.Biol.Chem 69, 435-441
3. Bernal, J. D., & Crowfoot, D. (1934). X-ray photographs of crystalline pepsin. Nature, 133(3369), 794-795.
4. Bernal, J. D., Fankuchen, I., & Perutz, M. (1938). An X-ray study of chymotrypsin and haemoglobin. Nature, 141(3568), 523-524.
5. Tanford, Charles; Reynolds, Jacqueline. Nature's Robots (Oxford Paperbacks) (p. 141-150). OUP Oxford. Kindle Edition.
6. Green, D. W., Ingram, V. M., & Perutz, M. F. (1954). The structure of haemoglobin-IV. Sign determination by the isomorphous replacement method. Proceedings of the Royal Society of London. Series A. Mathematical and Physical Sciences, 225(1162), 287-307.
7. Kendrew, J. C., Bodo, G., Dintzis, H. M., Parrish, R. G., Wyckoff, H., & Phillips, D. C. (1958). A three-dimensional model of the myoglobin molecule obtained by x-ray analysis. Nature, 181(4610), 662-666.
8. Kendrew JC (1961) The three‐dimensional structure of a protein molecule. Sci Am 205:96–110.
9. Perutz, M. F., Rossmann, M. G., Cullis, A. F., Muirhead, H., Will, G., & North, A. C. T. (1960). Structure of hæmoglobin: a three-dimensional Fourier synthesis at 5.5-Å. resolution, obtained by X-ray analysis. Nature, 185(4711), 416-422.













![[BioS 레터]무균주사제 공급망 변화와 CDMO 대응](https://img.etoday.co.kr/crop/77/77/2262816.jpg)


